Olá, pessoal, meu nome é Bruna, sou cientista de dados no Hospital Israelita Albert Einstein e, neste artigo, quero contar sobre um projeto muito bacana, do qual eu tenho bastante orgulho de participar, e que busca desenvolver soluções de inteligência artificial (IA) para serem utilizadas no Sistema Único de Saúde (SUS).
No Brasil, o SUS enfrenta inúmeros desafios na gestão de exames médicos, que são essenciais para o diagnóstico e tratamento dos pacientes. Atualmente, não existe uma plataforma que unifique e consolide todos os exames realizados, o que resulta na perda de informações valiosas ao longo do tempo. Sem um sistema centralizado, o histórico clínico do paciente pode extraviar-se, levando à repetição dos exames e ao desperdício de recursos já escassos na saúde pública.
Diante dessa realidade e do fato de que enfrentamos uma distribuição desigual de médicos especialistas em várias regiões do nosso país, o projeto PROADI-SUS Banco de Imagens surgiu com o objetivo de oferecer alternativas inovadoras para o armazenamento e compartilhamento de exames médicos no SUS. Por meio da tecnologia, incluindo o uso de IA, buscamos maximizar o uso dos dados clínicos, otimizando a continuidade e a qualidade do cuidado ao paciente, além de promover uma gestão mais sustentável dos recursos do sistema de saúde brasileiro. Neste artigo, vou explicar um pouco mais sobre esse projeto e como ele ajuda a levar tecnologia de ponta ao SUS.
Imagem gerada com Chat-GPT usando o prompt “Crie uma imagem representando a digitalização do Sistema Único de Saúde brasileiro”
O que é o projeto PROADI-SUS?
É o Programa de Apoio ao Desenvolvimento Institucional do Sistema Único de Saúde (PROADI-SUS), uma iniciativa que inclui os seis principais hospitais do Brasil e o Ministério da Saúde. Estabelecido em 2009, sua missão é fortalecer o SUS por meio de projetos focados em formação de recursos humanos, pesquisa, avaliação e integração de tecnologias, gestão e assistência especializada.
Um desses projetos é o Banco de Imagens Médicas, uma parceria entre o Hospital Israelita Albert Einstein e o Ministério da Saúde, cujo objetivo é o desenvolvimento de uma plataforma chamada Vendor Neutral Archive (VNA) para armazenamento e gerenciamento de imagens médicas do SUS em todo o país. A plataforma não apenas centraliza exames de imagem, mas também integra soluções baseadas em IA para dar suporte às decisões diagnósticas, garantindo que profissionais de saúde e pacientes de todas as regiões do Brasil tenham acesso a exames de alta qualidade, promovendo melhora no monitoramento de doenças e na avaliação de respostas ao tratamento dos pacientes.
Ao estimular o desenvolvimento e uso da IA, desejamos aumentar a precisão do diagnóstico, agilizar o processo de triagem dos pacientes e facilitar a detecção precoce de doenças, melhorando, em última análise, a saúde dos pacientes e reduzindo as desigualdades no acesso à saúde em todo o Brasil.
Mas quais soluções de IA são desenvolvidas pelo projeto?
No começo, desenvolvemos soluções baseadas em IA para dar suporte ao diagnóstico de três condições de alto impacto e relevância para o SUS: síndrome congênita causada pelo vírus Zika, melanoma cutâneo, e tuberculose pulmonar, com base em dados de tomografias computadorizadas de cabeça, fotos clínicas de melanoma e radiografias de tórax, respectivamente.
Recentemente, expandimos esses modelos para incluir outros casos de uso, mas, neste artigo, vou focar em apenas um dos modelos que temos e que, por coincidência, é o que eu ajudo a desenvolver. Trata-se de um algoritmo que segmenta e calcula automaticamente os volumes de estruturas cerebrais a partir de tomografias computadorizadas de cabeça. Inicialmente desenvolvemos o algoritmo para lidar com exames de pacientes com Zika, porém expandimos o seu uso para outras condições neurológicas, que também resultam em alterações nos volumes das estruturas cerebrais.
Antes de comentar um pouco mais sobre esse algoritmo, preciso falar sobre o tipo de dado que ele usa: a tomografia computadorizada.
O que é uma tomografia computadorizada?
A tomografia computadorizada, também chamada de TC, é uma técnica sofisticada de imagem médica baseada na utilização de raios-X para criar imagens tridimensionais altamente detalhadas das estruturas internas do corpo. Essa capacidade de obtenção de imagens em 3D torna a TC uma ferramenta essencial para o diagnóstico, planejamento cirúrgico e monitoramento de tratamentos. Em relação ao exame de ressonância magnética (RM), a TC possui menor tempo de aquisição, é mais barata e possui uma distribuição mais ampla e acessível no SUS.
A TC opera com base na emissão e captura de múltiplos feixes de raio-X emitidos em diferentes ângulos em torno do paciente. Essas projeções de raio-X são posteriormente combinadas por um computador para reconstruir imagens transversais, semelhante a “fatias” do corpo em intervalos milimétricos, também chamados de slices. O resultado é uma série de imagens em seção transversal que oferece uma visão detalhada das estruturas internas, incluindo ossos, órgãos, vasos sanguíneos e tecidos moles, permitindo, inclusive, a reconstrução tridimensional destas estruturas. Essa característica faz com que a TC seja considerada uma imagem volumétrica, composta não por pixels, mas sim por voxels.
Do ponto de vista computacional, as séries de imagens transversais são organizadas em arquivos no formato DICOM dentro de uma pasta. O formato DICOM (Digital Imaging and Communications in Medicine, ou em português, Comunicação de Imagens Digitais na Medicina) é um conjunto de normas que unifica o formato de exames de diagnóstico como TC, mamografia e RM no meio eletrônico. Seu principal objetivo é garantir que as imagens de diagnóstico obtidas em exames, mesmo quando realizadas em equipamentos de diferentes fabricantes, possam ser lidas por qualquer aparelho no formato digital.
Quando as séries de uma TC são armazenadas no formato DICOM, elas possuem duas informações importantes: a que está contida nos voxels da imagem adquirida e os metadados – que são informações relacionadas à imagem que está sendo adquirida: dados do paciente, do médico solicitante, do hospital no qual o exame foi realizado, por exemplo. Além de características da própria imagem, como número de slices, espaçamento entre os slices, coordenadas dos slices nos eixos x, y e z, e do equipamento utilizado, como a marca do tomógrafo e os parâmetros de configuração do aparelho.
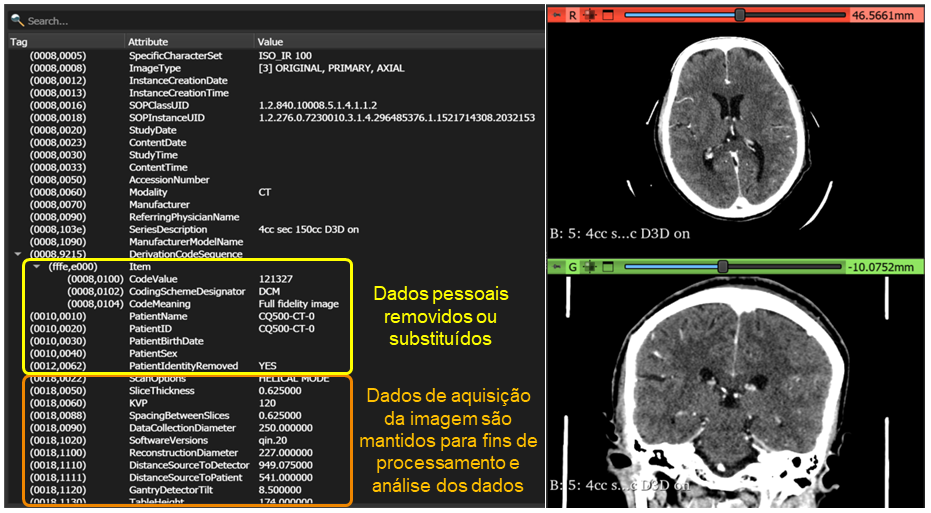
Exemplo de um arquivo DICOM de uma série de um exame. À esquerda, estão os metadados da imagem. Boa parte desses dados são removidos e/ou desidentificados para preservar a privacidade e atender as demandas da Lei Geral de Proteção de Dados (LGPD). À direita, vemos os dados dos voxels da imagem. Nessa figura, usamos como exemplo um exame disponível na internet, oriundo de um conjunto de dados chamado CQ500
Em um exame de TC é possível realizar a aquisição de uma ou mais séries, dependendo do protocolo realizado pela equipe da radiologia. Por exemplo, um exame pode conter séries adquiridas em diferentes momentos de administração de contraste na veia do paciente. A partir da estrutura de organização dos exames de TC, é possível observar que este tipo de exame possui uma complexidade de manuseio, dado que a quantidade de arquivos nos quais ele é armazenado é grande e, por consequência, exige uma grande quantidade de espaço de armazenamento computacional. Para fins ilustrativos, um exame de TC de cabeça, em média, ocupa 500MB de espaço e um conjunto de dados com cerca de mil exames de TC (o que é uma quantidade comum para um treinamento de um algoritmo de IA), ocupa 500GB de espaço em um volume de armazenamento de dados. A fim de lidar com estas questões de maneira mais eficiente, é comum realizarmos a conversão do formato DICOM para o formato NifTI, que é mais leve e fácil de manusear, pois trata-se de um único arquivo.
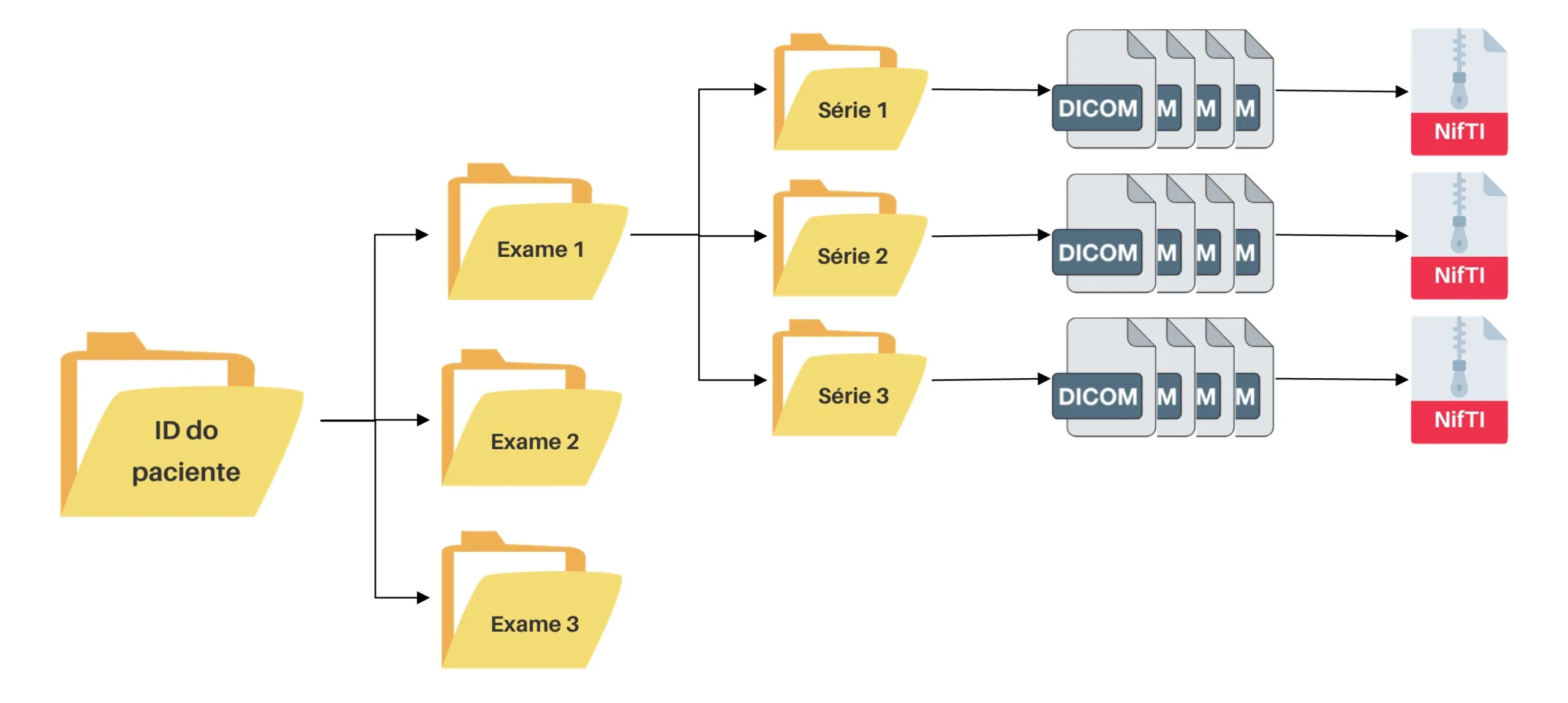
Estrutura de arquivos de exames de tomografia computadorizada. Um mesmo paciente pode realizar um ou mais exames, e cada um destes exames, por sua vez, pode ter uma ou mais séries cujos slices estão no formato DICOM. É comum a conversão destes arquivos para o formato NifTI, que compacta esses arquivos
Agora sim, vamos ao funcionamento do algoritmo
O algoritmo que desenvolvemos realiza uma tarefa de visão computacional bem conhecida: a segmentação de objetos. Aqui, minha sugestão é a leitura do excelente artigo da Maria Fernanda Souza sobre visão computacional, que explica direitinho essa e outras tarefas comuns na área. Mas, em resumo, o modelo delimita exatamente as estruturas intracraniana e dos ventrículos laterais (uma região do sistema nervoso central responsável por várias coisas, dentre elas, a produção do líquido encefalorraquidiano). Uma vez que conseguimos delimitar exatamente essas estruturas em todos os slices de um exame de TC de cabeça, conseguimos calcular o volume dessas estruturas.
Isso é muito interessante, pois na rotina clínica, o médico radiologista só sabe que uma estrutura cerebral aumentou ou diminuiu quando essa mudança é perceptível a olho nu. Mudanças mais sutis são difíceis de serem percebidas, porém seriam extremamente úteis de serem captadas, já que elas podem indicar a presença de alterações neurológicas nos pacientes, como lesões, tumores, hidrocefalia e outras alterações. O uso de um algoritmo de segmentação pode auxiliar na detecção precoce dessas condições, permitindo um tratamento rápido e evitando complicações futuras.
Aqui no projeto, nós construímos esse modelo baseado em uma arquitetura de redes neurais convolucionais. Vocês conhecem esse tipo de modelo?
Uma rede neural convolucional (ou CNN, do inglês, Convolutional Neural Network) é um tipo de modelo de IA inspirado na forma como o cérebro humano processa imagens. Ela é muito utilizada no campo da visão computacional porque consegue identificar padrões complexos em imagens de forma automática. Em vez de analisar cada voxel de uma imagem individualmente, as CNNs utilizam filtros (ou kernels) que percorrem a imagem e detectam características visuais, como bordas, texturas ou formas. Isso torna possível que a rede aprenda a reconhecer objetos ou distinguir padrões específicos, como rostos, animais ou até doenças em exames médicos.
O processo de uma CNN envolve várias camadas: as camadas de convolução extraem as características importantes da imagem; as camadas de pooling reduzem a quantidade de dados sem perder informações relevantes; e as camadas finais (normalmente de redes neurais tradicionais) fazem a predição da tarefa para a qual ela foi treinada. No nosso caso, a CNN classifica cada voxel do exame de TC de cabeça como pertencente ou não à região intracraniana e à região ventricular. A saída do modelo é um arquivo NifTI que contém essa classificação a nível de voxels e que chamamos de máscaras intracraniana e ventricular.
Usando um software gratuito de análises de imagens médicas, chamado 3D Slicer, nós conseguimos visualizar esse resultado sobrepondo essas máscaras ao exame de TC original. É o que a gente pode observar na a seguir.
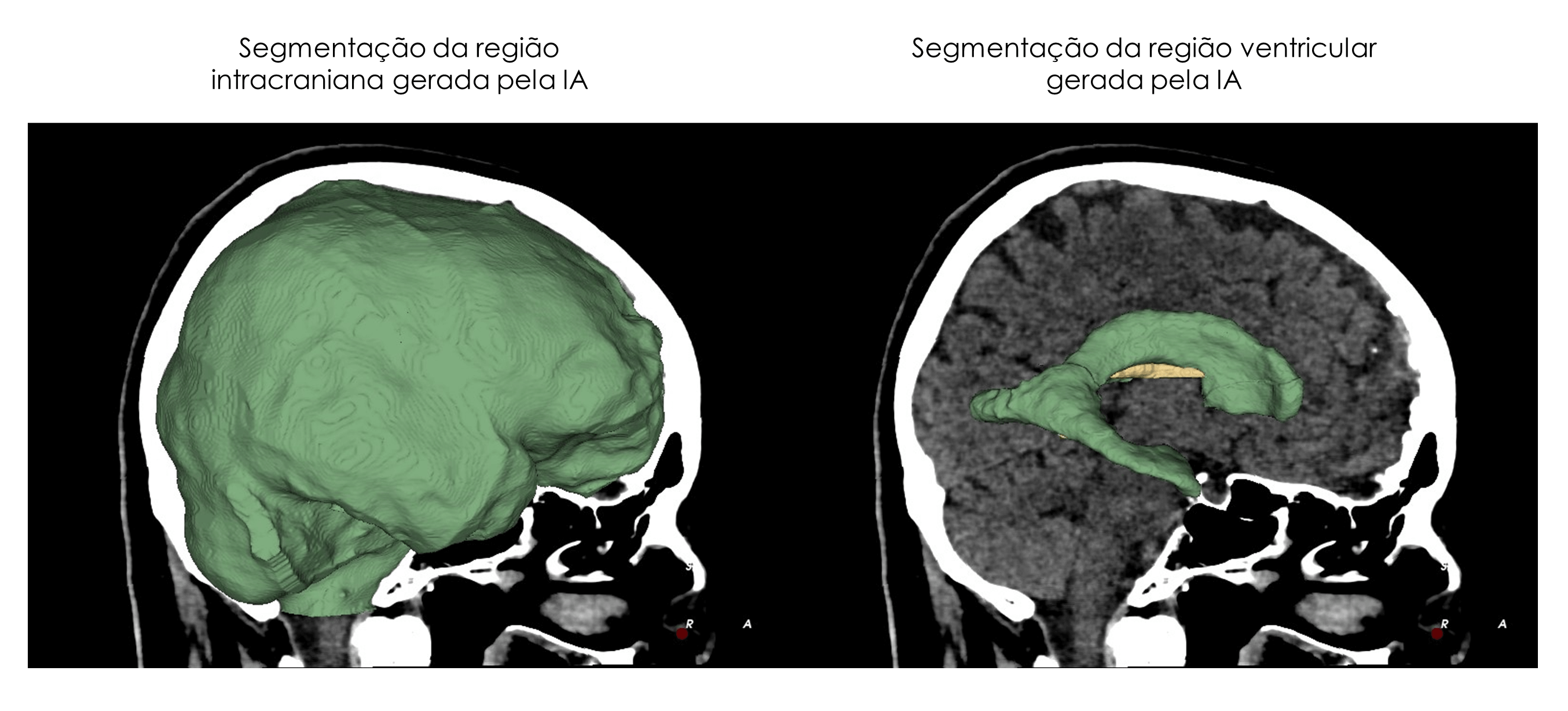
Exemplo de saída do algoritmo de segmentação das regiões intracraniana e ventricular obtidas a partir de um exame de TC de cabeça
Como sabemos que o modelo executou bem esta tarefa?
Nós comparamos as máscaras que ele gera com as máscaras geradas manualmente com o uso do 3D Slicer por médicos especialistas. Há algumas métricas que usamos para avaliar quantitativamente esses resultados. O coeficiente de similaridade DICE e a distância de Hausdorff são duas delas.
O coeficiente DICE é uma medida de similaridade entre duas máscaras (no nosso caso, a máscara predita pelo modelo e a máscara gerada pelo médico especialista). Ela varia de 0 a 1 e, quanto mais próxima de 1, melhor. Já a distância de Hausdorff mede a distância entre duas máscaras e possui um valor positivo, porém quanto mais próximo de 0, melhor, pois indica que a distância entre a máscara que o modelo gerou e a máscara verdadeira gerada pelo médico, estão próximas.
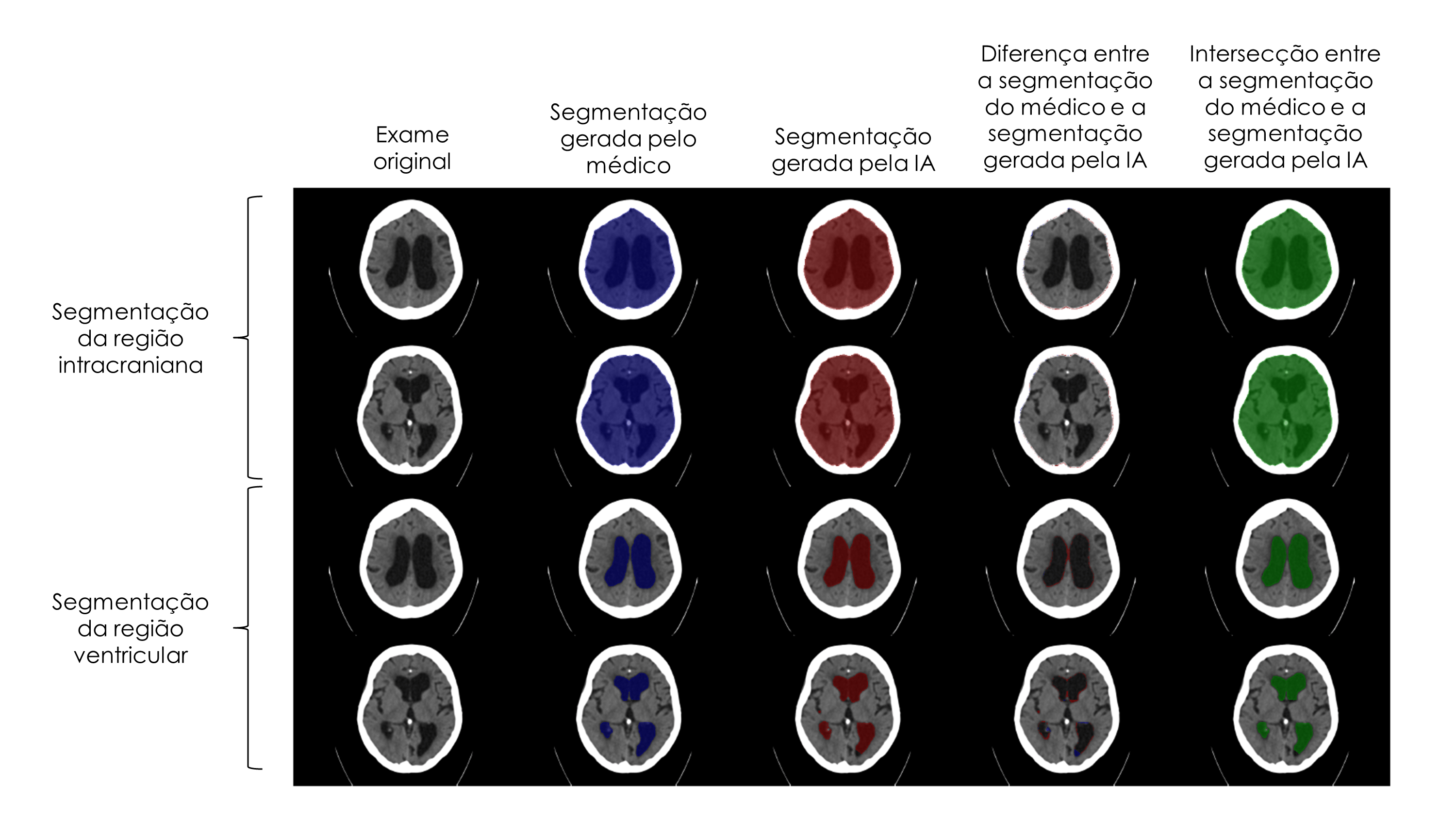
Comparação das máscaras intracraniana e ventricular geradas manualmente pelo médico especialista (segunda coluna) e pelo modelo de IA (terceira coluna). É possível fazer a subtração das máscaras para saber quais regiões da imagem o modelo deixou de considerar como parte das regiões intracraniana e ventricular ou as regiões que ele considerou como parte delas, mas que o médico especialista não considera (quarta coluna). Por fim, também é possível observar as regiões nas quais as máscaras da IA e do médico se sobrepuseram (quinta coluna). Este é um exemplo de imagem na qual o modelo performou bem a tarefa, pois quase não é possível observar diferenças na quarta coluna e na quinta coluna, há uma grande sobreposição entre as máscaras
Como garantir a eficácia e a segurança desse e dos outros algoritmos?
Aqui no projeto construímos soluções de IA com bases em exames médicos reais de pacientes de diferentes origens, regiões, sexos, idades e cujos exames tenham sido adquiridos a partir de diferentes aparelhos de imagem médica, como tomógrafos de diferentes marcas e com diferentes tempos de uso (mais velhos ou mais novos) para refletir a variabilidade dos exames feito pelo SUS. Também realizamos a avaliação da performance dos nossos modelos de acordo com todos esses parâmetros que mencionei, sempre de forma anonimizada, para garantir que o modelo tenha um bom desempenho em todas essas condições. Dessa forma, desenvolvemos ferramentas de IA que além de serem eficazes, também são equitativas, transparentes e respeitosas à privacidade do paciente.
Como saber mais sobre o assunto?
O tema de inteligência artificial na saúde, particularmente em imagens médicas, possui um conteúdo que ainda é majoritariamente produzido na língua inglesa. Minha dica, para quem tem dificuldade no idioma, é que vocês usem as ferramentas de IA disponíveis na internet para conseguir entender esses conteúdos. Eu faço isso boa parte do tempo. Além disso, aqui no Brasil temos um congresso na área de radiologia bem grande e importante, chamado Jornada Paulista de Radiologia (JPR). Nos últimos anos, os organizadores têm lançado desafios de imagens médicas com dados brasileiros na plataforma de desafios de ciência de dados do Kaggle. Participar desses e de outros desafios na área é uma excelente oportunidade para aprender, na prática, vários dos conceitos abordados neste artigo. Minha sugestão é que vocês acompanhem os fóruns de discussão e testem os códigos que os participantes compartilham por lá. Faço isso e sempre aprendo muito.
Dicas de materiais
- Curso sobre deep learning. É pago, mas dá para pedir auxílio financeiro (eles isentam o pagamento do curso). Podem pedir ajuda para o Chat-GPT para escrever o pedido de auxílio que dá super certo.
- Canal do YouTube de um pesquisador que trabalha com imagens médicas.
- Desafio deste ano do Congresso JPR. O objetivo era predizer a idade biológica dos pacientes com base em exames de TC de cabeça. Uma equipe do meu projeto ficou em 5º lugar nela.
Autora Bruna Garbes Gonçalves Pinto é cientista de dados no Hospital Albert Einstein, atua no projeto PROADI-SUS onde desenvolve algoritmos de deep learning para o auxilio na interpretação de imagens médicas. Bruna é formada em ciência e tecnologia pela Universidade Federal do ABC e mestre em bionformática pelo Instituto de Matemática e Estatística da USP. Revisora Jayne L. Oliveira, jornalista e produtora editorial.
Este conteúdo faz parte do PrograMaria Sprint IA e Dados.

